Hei! Jeg heter Martine og sammen med André er jeg i praksis hos Gades laboratorium for patologi, som tilhører KI1 på Haukeland Universitetssykehus. Vårt prosjekt dette semesteret er å teste ut om man kan bruke kunstig intelligens for å kvantifisere mitokondrier i kreftceller, i tillegg til konvensjonelle kjemiske kvantifiseringsmetoder som brukes på lab i dag.
Kreftcellene er dyrket på lab, og tilført «friske» mitokondrier. Dette er knyttet opp mot et forskningsprosjekt som vil undersøke hvordan kreftceller utnytter de tilførte mitokondriene for egen energiproduksjon. Det var veldig nyttig å ha hatt FARM280 for å forstå de mange fremmedordene patologene vi samarbeider med bruker!
Oppgaven vår var ganske åpen i begynnelsen, og vi fikk komme med forslag til fremgangsmåter og prosedyrer selv. Det var deilig å få tillit fra start, og siden da har vi stort sett jobbet selvstendig. Samtidig hadde vi vært helt ute å sykle uten god hjelp og svar på våre mange spørsmål, både fra patologene og fra ingeniørene på MIC (Molecular Imaging Centre) der vi stort sett sitter og jobber.
Vi har landet på å bruke en programvare som heter Imaris. Imaris kan analysere 3D-bilder tatt av mikroskoper. I Imaris begynner vi med et stort bilde som dette:
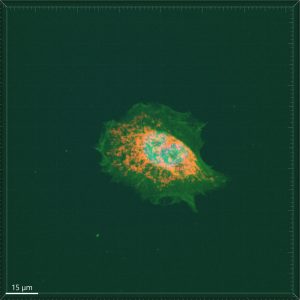
Bildet er tatt ved å markere ulike komponenter i cellen med ulike markører slik at de blir fluorescerte, og dermed sender ut lys når de bestråles med laser. Dette har vi fått være med å prøve! Det grønne er selve cellen, mens det rød er fra mitokondrier. Etter vi har behandlet bildet i Imaris ser det ut noe sånt som dette:
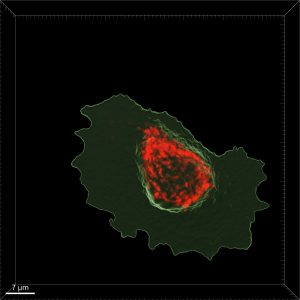
Det er ikke selve visualiseringen som er interessant, men heller at Imaris gir statistikk på visualiseringene vi har gjort. Vi henter blant annet ut volumet til hver celle og beregner hvor stor prosentandel av hver celle som er mitokondrier. Så langt i semesteret har vi kun jobbet med datainnsamling, noe jeg har fått større respekt for enn jeg hadde da jeg tok INF161 hvor vi fikk dataen ferdig innsamlet. Vi har så langt katalogisert ca. 40 celler, og vil ha minst 150 celler før vi går videre med prosjektet vårt. Med andre ord har vi fremdeles mye å gjøre!