Hei igjen!
Nå nærmer det seg dessverre snart slutten på mitt praksisopphold hos Patologi i Vest (PiV) på Eitri. Jeg har lært utrolig mye og trives veldig godt her. Jeg kommer til å savne både stedet og menneskene som jobber her når perioden er over.
Siden sist har jeg jobbet videre med prosjektet mitt; hatt noen suksesser, men også støtt på flere problemer som jeg har jobbet med å løse. I tillegg har jeg fått vært en hel dag på histologilaboratoriet på patologiavdelingen, vært med på en del møter og deltatt og presentert på prosjektmøte for hele Patologi i Vest, med deltakere fra både Stavanger, Førde, Haugesund og Bergen.
Da jeg besøkte histologilaboratoriet fikk jeg en omvisning som varte hele dagen, hvor jeg fikk være ca. 30-45 min på hver «post»; mottak, makro uttak, prosessering, støpning i parafin, snitting, farging, scanning/utdeling av snitt. Dette var veldig lærerikt, og jeg er veldig takknemlig for denne muligheten. Jeg har tidligere vært på besøk hos nyrelaboratoriet, men det var likevel veldig mye nytt å lære på histologilaboratoriet ettersom det er i en mye større skala; flere digitale snitt blir produsert, mer automatiserte prosesser og mange flere ansatte. Bildet under viser maskinen som farger snittene automatisk.

Målet med prosjektet mitt er å analysere variasjon av hematoxylin-eosin (HE) farging i digitale snitt fra nyrebiopsier og dedektere avvik fra optimal farging. Da jeg publiserte forrige blogginnlegg var status i prosjektet mitt at jeg så vidt hadde begynt å eksperimentere med clustering av bildene. På det tidspunktet tok jeg kun utgangspunkt i et lite utsnitt av hvert bilde, en såkalt «image patch» (som jpeg format) og brukte en kombinasjon av verdier kalkulert av QuPath og Python. Å eksportere bildene som jpeg viste seg å være en dårlig idé ettersom fargeverdiene endret seg på grunn av bildekompresjon. I tillegg ga det ikke helt mening å kombinere fargeverdier fra QuPath og de jeg kalkulerer i Python, ettersom jeg ikke har noe informasjon om hvordan verdiene i QuPath blir kalkulert. Ettersom QuPath kun gir meg gjennomsnitt, median og standardavvik har jeg gått over til å kun gjøre bildeanalyse i python. Jeg har nå gått over til å eksportere bildende i tif format, et format uten kompresjon, slik at bildeverdiene ikke ble forandret. I tillegg tar jeg nå utgangsspunkt i hele digitale snitt. Bildet under viser et eksempel av et eksportert bilde og tilsvarende maske for det bildet.
Som en del av analysen i python har jeg arbeidet en del med å lage en funksjon som maskerer bort bakgrunnen. På den måten kan jeg kalkulere fargeverdier, uten at de hvite pikslene inkluderes. Videre gjorde jeg fargeanalyse og plottet verdiene fra ulike fargerom. Deretter regnet jeg ut statistiske verdier (gjennomsnitt, median, entropi, skjevhet, standardavvik osv..) Bildet under viser hue, saturation og intensity for et spesifikt bilde, med og uten maske. Man kan se at de pikselverdiene som er hvite ikke inkluderes i plottene hvor masken er brukt.

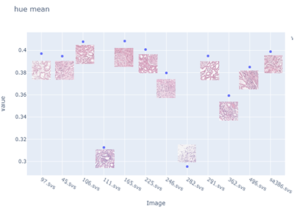
Bildet under viser gjennomsnittlig hue verdi for de ulike bildene plottet mot hverandre. Man kan se at det er to bilder som skiller seg ut fra resten. Dette har vist seg å være de to bildene med optimal farging.
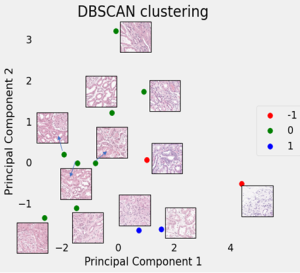
Disse verdiene ble satt sammen i et datasett. Jeg reduserte så antall dimensjoner til 2 (ved bruk av PCA), slik at det ble lettere å visualusere clustering av dataen. Jeg eksperimenterte med ulike typer clustering. Density-based clustering, som lager clusters basert på tettheten av punkter, markerer datapunkter som utliggere dersom de ikke passer inn i en cluster. Jeg hadde håpet at utliggerne ville være de bildene med dårlig fargekvalitet. Da jeg fikk vite hvilke bilder som faktisk hadde ønsket farging viste det seg at dette kun gjaldt to av bildene, og at det var disse som ble markert som utliggere. Dermed funker ikke denne metoden for det gjeldende datasettet. Bildet under illustrerer metoden som ikke virket.
Planen videre nå er å utvide datasettet til å inneholde flere bilder med ønsket farging, slik at datasettet får en bedre balanse. Deretter skal bildene deles opp i mindre deler for å simulere mer data. Det gjøres dermed kalkulering av fargeverdier på de ulike datapunktene som igjen brukes til clustering. Håpet er da at datapunktene som tilhører samme bildet havner sammen og at utsnittene med dårlig farging blir utliggere. Ettersom det nå er kjent hvilke bilder som har bra farging er det også et mål å finne frem til de statistiske verdiene som korrelerer best med bildenes merkelapp (hvor bra fargingen i bildet er). Da har man muligheten til å definere terskelverdier for ulike statistiske verdier for å klassifisere optimal farging og ikke optimal farging.
Jeg er veldig glad for at praksis har blitt inkorporert i studieplanen for medisinsk teknologi på UiB. Gjennom praksisperioden lærer man mye nytt, samtidig som man får mulighet til å bruke teori som man har lært i praksis. Et eksempel på dette i mitt tilfelle er at jeg tar INF264 (introduksjon til maskinlæring) parallelt med MTEK200 (praksis). Dette har gjort at jeg har kunnet bruke teori om PCA og clustering, som jeg har lært i forelesningene, i praksis, noe som har gitt meg økt læringsutbytte. Siden medisinsk teknologi er et veldig bredt felt kan man også tilegne seg kunnskap som ikke dekkes av pensum på studiet, i mitt tilfelle patologi. Gjennom praksisperioden hos Patologi i Vest har jeg fått ny motivasjon og oppdaget at jeg har en stor interesse for maskinlæring i forbindelse med medisinsk bildeanalyse.
Jeg er veldig takknemlig for måten praksisplassen min har tatt imot meg; jeg har blitt inkludert i arbeidsmiljøet og blitt gitt gode utfordringer.