Hei igjen! Thea og jeg er fortsatt i praksis hos Patologi i Vest (PiV), og vi jobber videre med prosjektet vårt! Prosjektet vårt går ut på å lage en automatisk analyse strategi for å vurdere kvaliteten på fargingen av nyrebiopsier. Spesifikt ser vi på farging av ikke-neoplastiske nyrebiopsier som er farget med immunhistokjemi (IHC). Her ønsker vi å se på dag-til-dag variasjon på fargekvaliteten ved hjelp QuPath, et open-source program for å se på biopsier, og Python.
Siden sist har vi jobbet mye med celledeteksjonen og klassifiseringen av plasmacellene i QuPath. Celledeteksjonene blir gjort med en utvidelse til QuPath som vi har tilpasset, mens den automatiske klassifisering har vi laget selv. Her har vi definert tre forskjellige kategorier; positive, negative og annet. De «positive» er plasmaceller som har blitt farget brune av IHC, de «negative» er plasmaceller og andre celler som ikke har blitt farget og «annet» er alle andre celledeteksjoner som ikke er korrekte, blant annet artefakter.
For å trene opp denne automatiske klassifiseringsmodellen måtte vi lage en «ground truth», celledeteksjoner som har blitt korrekt klassifisert av oss. Nedenfor kan du se vår «ground truth» til venstre og klassifiseringsmodellen sin prediksjon til høyre. Det er noe variasjon mellom dem, hovedsakelig hvor modellen vår klassifiserer for mange deteksjoner som «annet».
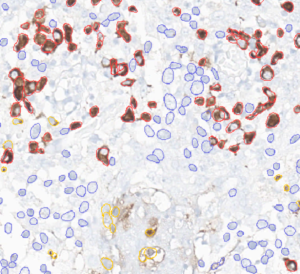
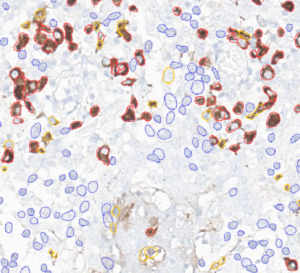
Vi har også begynt å ta inn dette til Python, hvor vi nå prøver å sjekke hvor nøyaktig modellen vår er. Videre skal vi også gjøre analyse på fargingen til deteksjonene som er kategorisert som positive. Det kommer til å bli veldig spennende å se hva vi kan få til med det, og det er veldig morsomt å se at vi får tatt i bruk det vi har lært i f.eks. INF161.