Denne hausten har Anne Mari og eg, Martine, fått praksisplass hos forskingsgruppa PIV på Eitri, i tilknyting til Haukeland Universitetssjukehus. Her deltek vi på eit prosjekt, som handlar om å forstå meir av det som skjer i nyrebiopsiar. Nærare bestemt ser vi på tubuli, dei små røyrstrukturane i nyrene. Dei har ansvar for å transportere væske, næringsstoff og avfall.
Kronisk nyresjukdom er eit alvorleg og vanleg helseproblem, og for å stille rett diagnose må patologar ofte gå gjennom biopsiar for å leita etter forandringar i nyrevevet. Det finst allereie gode system for å vurdere blodårer og glomeruli, men tubuli har blitt litt oversette. Her er det mykje, som framleis er uklart, og det er akkurat det vi prøver å bidra til å kartleggje!
Vi er nett ferdig med første delen av prosjektet, der vi har ekstrahere tubuliene. Vi har tatt utgangspunkt i tubuli som er annotert og klassifisert i QuPath, eit dataprogram for digital patologi. Du kan sjå eit slikt QuPath snitt på bilete, med ulike tubuli med stor variasjon i form og farge (indikerer annotasjon).
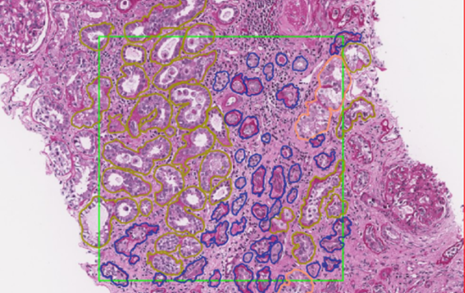
Videre har vi skrive vår eigen kode i Python, for å klippe ut alle desse tubuliene, basert på koordinatane. Resultatet er ei samling av små bileteutsnitt, som vi kan jobbe vidare med. Det har blitt ein del prøving og feiling, debugging, og eit par «aha-opplevingar» når koden endeleg køyrde slik vi ville. Vi har valt å klippe ut utsnitta på ulike måtar. Noen er sentrerte og i noen er bakgrunnen maskert. Målet med dette er å kunne finne ut kva som vil være det beste formatet, for vidare bruk.
Neste steg i prosjektet blir å hente ut eigenskapar frå alle tubulus-bileta. Dette skal vi gjøre ved hjelp av deep learning. Deretter skal vi evaluere desse eigenskapane og utforske ulike strategiar for clustering, altså korleis bileta kan grupperast automatisk. Til slutt skal vi vurdere om resultata faktisk gir meining i ein klinisk samanheng.
Vi gler oss til å lære meir, og ikkje minst til å sjå korleis resultata våre kan brukast i praksis.
Vi snakkast i neste blogginnlegg.